Machine Learning with Scikit Learn (Part II)
Machine Learning with Scikit Learn (Part I)(2015/8/10)のつづき。今回は、後編のPartIIの動画の内容を簡単にまとめたい。
4.1 Cross Validation
ラベル付きデータが少ないときに有効な評価法であるK-fold cross-validationについての説明。訓練データをK個のサブセットに分割し、そのうち1つのサブセットをテストデータに残りK-1個のサブセットを訓練データにして評価する。これをテストデータを入れ替えながらK回評価し、その平均を求める。
この手順は下のように書ける。bool型のマスクを使ってテストデータと訓練データをわけている。
k = 5 n_samples = len(X) fold_size = n_samples // k scores = [] masks = [] for fold in range(k): test_mask = np.zeros(n_samples, dtype=bool) test_mask[fold * fold_size:(fold+1)*fold_size] = True X_test, y_test = X[test_mask], y[test_mask] X_train, y_train = X[~test_mask], y[~test_mask] classifier.fit(X_train, y_train) scores.append(classifier.score(X_test, y_test)) print np.mean(scores)
sklearn.cross_validationモジュールを使うと上と同じことが3行で書ける。
from sklearn.cross_validation import cross_val_score scores = cross_val_score(classifier, X, y, cv=5) print np.mean(scores)
データの分割方法は、StratifiedKFold、KFold、ShuffleSplit、LeaveOneOutなどが用意されている。たとえば、StratifiedKFoldのデータの分割方法は、
cv = StratifiedKFold(iris.target, n_folds=5) for train, test in cv: print(test)
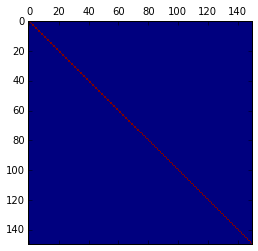
で確かめられる。可視化してみると

となる。行がCross Validationの各fold、列がirisの150個のデータである。青が訓練データ、赤がテストデータを意味している。irisデータは最初の50個がクラス1、次の50個がクラス2、次の50個がクラス3なので、各foldのテストデータはクラスの比率を維持したまま選ばれていることがわかる。
一方、単純なKFoldだと

と単純に等分にされる。データをシャッフルしないと各foldの正解ラベルが偏ってしまい、精度が大きくばらつくことになる。データをシャッフルするにはKFoldの引数にshuffle=Trueを入れておけばOK。
テストデータの選択をランダムで行うShuffleSplitもある。

ラベル付きデータ数が非常に少なく、K個のfoldに分割もできない場合は、テストデータとして1サンプルだけ選び、残りサンプルすべてを訓練データにするLeaveOneOutが使える。
cross_val_score()のデフォルトでは、StratifiedKFoldが使われるが、cvオプションに上記の任意のオブジェクトを指定できる。例えば、テストデータがランダムに選択されるShuffleSplitを使ってCross Validationをしたければ下のように書く。
cv = ShuffleSplit(len(iris.target), n_iter=5, test_size=.2) cross_val_score(classifier, X, y, cv=cv)
4.2 Model Complexity and GridSearchCV
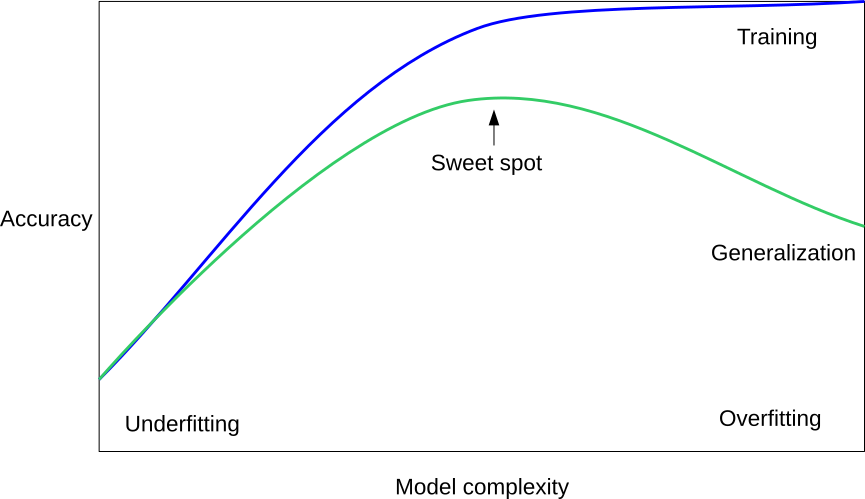
モデルのOverfitting/Underfittingとハイパーパラメータを最適化するグリッドサーチについての説明。通常の機械学習モデルは下のような関係が成り立つ。
 引用:amueller/scipy_2015_sklearn_tutorial · GitHub
引用:amueller/scipy_2015_sklearn_tutorial · GitHub
モデルが単純すぎるとUnderfittingぎみになり、訓練データとテストデータの両方とも精度が低い。一方、モデルを複雑化するとOverfittingぎみになり、訓練データの精度はすごく高くなるが、テストデータの精度が逆にすごく悪くなる。もっとも汎化された望ましいモデルは、テストデータの精度がもっとも高くなるSweet Spotの場所になる。
説明ではK近傍法の例が挙げられている。K近傍法のハイパーパラメータは近傍数のn_neighbors(K近傍法のKと同じ)である。このハイパーパラメータを変えて学習すると下のようになる。
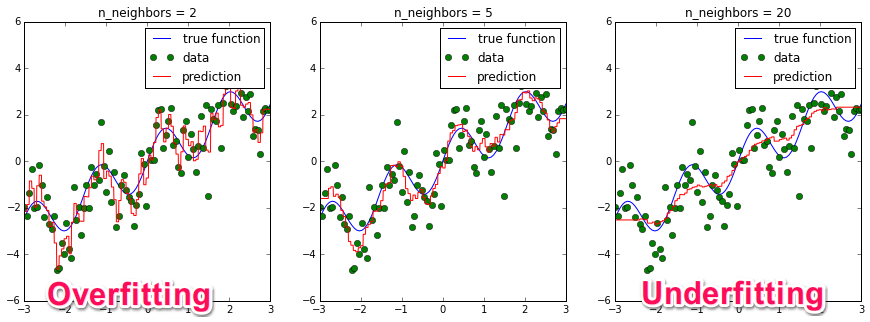
一番左のK=2はモデルが複雑で訓練データの予測精度は高いがテストデータの精度は低いためOverfittingぎみ。一方、一番右のK=20はモデルが単純で訓練データに対してもテストデータに対しても精度は低いためUnderfittingぎみと解釈できる。真ん中のK=5が最適モデルとなる。
このような最適モデルを探す一般的な方法はなく、モデルのハイパーパラメータの組み合わせを力づくで評価して見つけるしかない。この最適なハイパーパラメータの探索にもCross Validationが使える。
from sklearn.cross_validation import cross_val_score, KFold from sklearn.neighbors import KNeighborsRegressor # generate toy dataset: x = np.linspace(-3, 3, 100) rng = np.random.RandomState(42) y = np.sin(4 * x) + x + rng.normal(size=len(x)) X = x[:, np.newaxis] cv = KFold(n=len(x), shuffle=True) # for each parameter setting do cross_validation: for n_neighbors in [1, 2, 3, 5, 10, 20]: scores = cross_val_score(KNeighborsRegressor(n_neighbors=n_neighbors), X, y, cv=cv) print "n_neighbors: %d, average score: %f" % (n_neighbors, np.mean(scores))
実行結果は、
n_neighbors: 1, average score: 0.586909 n_neighbors: 2, average score: 0.706250 n_neighbors: 3, average score: 0.738065 n_neighbors: 5, average score: 0.769667 n_neighbors: 10, average score: 0.740026 n_neighbors: 20, average score: 0.639899
となり、先のグラフからもわかるようにn_neighbors=5の平均精度がもっともよいことがわかる。validation_curve()を使うと横軸にハイパーパラメータ、縦軸に精度のグラフが簡単に描ける。
from sklearn.learning_curve import validation_curve n_neighbors = [1, 2, 3, 5, 10, 20] train_scores, test_scores = validation_curve(KNeighborsRegressor(), X, y, param_name="n_neighbors", param_range=n_neighbors, cv=cv) plt.plot(n_neighbors, train_scores.mean(axis=1), label="train score") plt.plot(n_neighbors, test_scores.mean(axis=1), label="test score") plt.legend(loc="best")
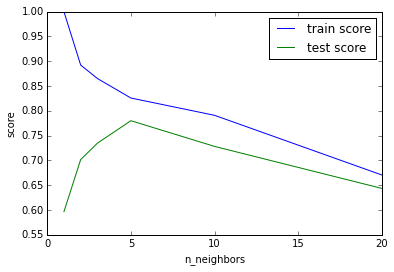
このグラフからもn_neighbors=5でテストデータの精度が最大化されることがわかる。
K近傍法はハイパーパラメータが1つしかないが、複数ある場合も同じように探索できる。たとえば、SVMはCとgammaという2つのパラメータがあるため二重ループでパラメータのすべての組み合わせを評価する必要がある。
from sklearn.cross_validation import cross_val_score, KFold from sklearn.svm import SVR for C in [0.001, 0.01, 0.1, 1, 10]: for gamma in [0.001, 0.01, 0.1, 1]: scores = cross_val_score(SVR(C=C, gamma=gamma), X, y, cv=cv) print "C: %f, gamma: %f, average score: %f" % (C, gamma, np.mean(scores))
実行結果は、
C: 0.001000, gamma: 0.001000, average score: -0.006242 C: 0.001000, gamma: 0.010000, average score: -0.004179 C: 0.001000, gamma: 0.100000, average score: 0.004933 C: 0.001000, gamma: 1.000000, average score: 0.003641 C: 0.010000, gamma: 0.001000, average score: -0.003968 C: 0.010000, gamma: 0.010000, average score: 0.016451 C: 0.010000, gamma: 0.100000, average score: 0.098317 C: 0.010000, gamma: 1.000000, average score: 0.088333 C: 0.100000, gamma: 0.001000, average score: 0.018481 C: 0.100000, gamma: 0.010000, average score: 0.180360 C: 0.100000, gamma: 0.100000, average score: 0.526633 C: 0.100000, gamma: 1.000000, average score: 0.498146 C: 1.000000, gamma: 0.001000, average score: 0.194592 C: 1.000000, gamma: 0.010000, average score: 0.596778 C: 1.000000, gamma: 0.100000, average score: 0.672521 C: 1.000000, gamma: 1.000000, average score: 0.724113 C: 10.000000, gamma: 0.001000, average score: 0.593179 C: 10.000000, gamma: 0.010000, average score: 0.622648 C: 10.000000, gamma: 0.100000, average score: 0.669068 C: 10.000000, gamma: 1.000000, average score: 0.772627
となり、C=10とgamma=1.0のスコアが0.77と最大であることがわかる。
このようなハイパーパラメータの探索は、グリッドサーチと呼ばれる。scikit-learnではグリッドサーチを簡単に行うGridSearchCVが提供されている。
from sklearn.grid_search import GridSearchCV param_grid = {'C': [0.001, 0.01, 0.1, 1, 10], 'gamma': [0.001, 0.01, 0.1, 1]} grid = GridSearchCV(SVR(), param_grid=param_grid, cv=cv, verbose=3)
パラメータの組み合わせは辞書で与える。戻り値のgridに対してデータをfit()させるとハイパーパラメータの学習とモデル学習が順番に実行される。。
grid.fit(X, y)
最初に与えたデータを使ってCross Validationによる最適なハイパーパラメータ探索が行われ、その後に与えたデータすべてを使ってモデル学習が行われる。Cross Validationで見つかった最適なパラメータと最適な精度は下のコードで取得できる。
print grid.best_params_ print grid.best_score_
{'C': 10, 'gamma': 1}
0.772226773354
全体の手順をまとめると
- 訓練データとテストデータに分割する
- 訓練データを使ってCross Validationでモデルの最適なハイパーパラメータを見つける
- 訓練データを使ってモデルを学習する
- テストデータで精度を評価する
2と3はfit()でまとめて行われる。絵で描くと
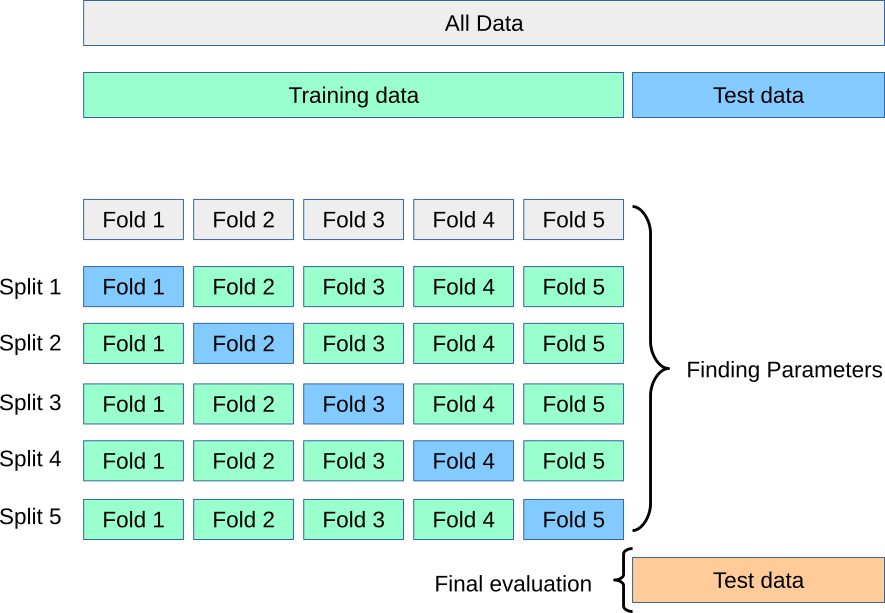 引用:amueller/scipy_2015_sklearn_tutorial · GitHub
引用:amueller/scipy_2015_sklearn_tutorial · GitHub
コードで書くと
from sklearn.cross_validation import train_test_split X_train, X_test, y_train, y_test = train_test_split(X, y, random_state=0) param_grid = {'C': [0.001, 0.01, 0.1, 1, 10], 'gamma': [0.001, 0.01, 0.1, 1]} cv = KFold(n=len(X_train), n_folds=10, shuffle=True) grid = GridSearchCV(SVR(), param_grid=param_grid, cv=cv) grid.fit(X_train, y_train) grid.score(X_test, y_test)
4.3 Analyzing Model Capacity
使っているモデルがOverfittingなのかUnderfittingなのかを見極めるためには学習曲線(learning curve)を描くとよい。学習曲線は、横軸に訓練サンプル数、縦軸に精度(スコア)を描いたグラフである。scikit-learnでは、learning_curveメソッドで簡単に描ける。例では、SVMのカーネルにlinear、poly、rbfを使った場合の学習曲線を比較している。モデルの複雑さは、linear < poly < rbfとなる。
まずは、SVMのカーネルにlinearを使った場合。
from sklearn.learning_curve import learning_curve from sklearn.svm import SVR training_sizes, train_scores, test_scores = learning_curve(SVR(kernel='linear'), X, y, cv=10, scoring="mean_squared_error", train_sizes=[0.6, 0.7, 0.8, 0.9, 1.0]) plt.plot(training_sizes, train_scores.mean(axis=1), label="training scores") plt.plot(training_sizes, test_scores.mean(axis=1), label="test scores") plt.legend(loc="best")
学習曲線のスコアにmean squared errorを使うと戻り値はNegative MSEが返るので注意。MSEは小さいほうが良いがスコアは大きいほうがよいため符号を逆にして返す仕様らしい。learning_curve()の戻り値は訓練データ数のリスト、各訓練データ数で学習したときの訓練スコア、テストスコアとなる。10-fold Cross Validationで評価しているため訓練スコアとテストスコアの両方がまとめて求められる。
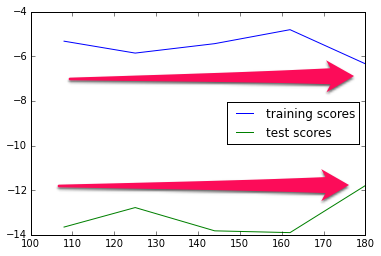
この学習曲線を見ると、訓練データ数を増やしても訓練スコア、テストスコアともに改善しない傾向があることがわかる。これはUnderfittingを意味する。つまり、そもそものモデルの表現力が貧弱なので頑張って訓練データ数を増やしても精度は改善しない。Underfittingなモデルに対しては、
- より複雑なモデルを使う(kernelをpolyやrbfにするなど)
- サンプルの特徴量を増やす
- 正則化を緩める(SVMのパラメータC)
などの対応を取るとよい。
次に、SVMのカーネルにrbfを使った場合。先のコードでkernel='rbf'に置き換えるだけ。
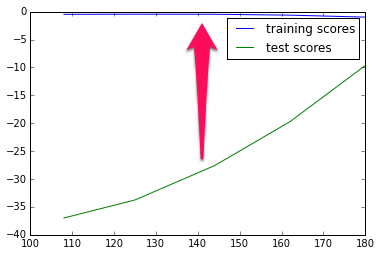
テストスコアに比べて訓練スコアが圧倒的に高い傾向がある。これはOverfittingを意味する。つまり、モデルが訓練データにフィットしすぎているため訓練データの精度は非常によくなるが、逆に汎化性能がなくなるためテストスコアが悪くなる。Overfittingなモデルに対しては、
- 特徴量を減らす
- より単純なモデルを使う
- 訓練データ数を増やす
- 正則化を強める
などの対応を取るとよい。
最後に、SVMのカーネルにpolyを使った場合。先のコードでkernel='poly'に置き換えるだけ。
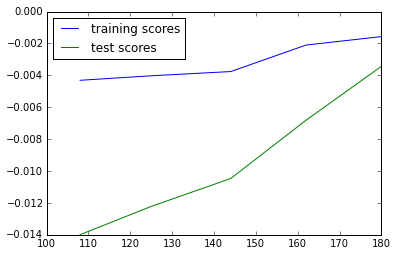
訓練データ数を増やすと訓練スコアとテストスコアの精度の差が小さくなり、収束することがわかる(スコア軸のスケールが前の2つに比べて非常に小さいことに注目)。このような学習曲線が得られるとよいモデルであると判断できる。つまり、今回のデータではkernel=polyを使うのが最適ということがわかる。
4.4 Model Evaluation and Scoring Metrics
モデルのさまざまな評価法についての説明が続く。分類モデルのscore()はデフォルトでは精度(accuracy)、正解したラベルの割合を返す。
from sklearn.datasets import load_digits from sklearn.cross_validation import train_test_split from sklearn.svm import LinearSVC digits = load_digits() X, y = digits.data, digits.target X_train, X_test, y_train, y_test = train_test_split(X, y) classifier = LinearSVC().fit(X_train, y_train) y_test_pred = classifier.predict(X_test) print "Accuracy: %f" % classifier.score(X_test, y_test)
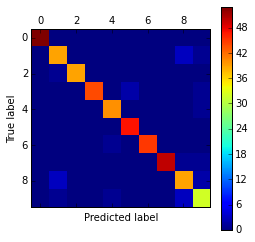
精度以外にもさまざまな評価法があり、sklearn.metricsに実装されている。多クラスの分類問題で有効な評価法が混同行列(confusion matrix)である。各クラスのサンプルがどんな間違われ方をしたのか直感的に把握できる。
from sklearn.metrics import confusion_matrix confusion_matrix(y_test, y_test_pred)
[[53 0 0 0 0 0 0 0 0 0] [ 0 39 0 0 0 0 0 0 3 1] [ 0 1 39 0 0 0 0 0 0 0] [ 0 0 0 44 0 2 0 0 0 1] [ 0 0 0 0 40 0 0 0 0 1] [ 0 0 0 0 0 47 0 0 0 0] [ 0 0 0 0 1 0 45 0 0 0] [ 0 0 0 0 0 0 0 50 1 1] [ 0 3 0 0 0 0 0 0 39 2] [ 0 1 0 0 1 0 0 0 3 32]]
グラフで可視化するとより直感的にわかる。
plt.matshow(confusion_matrix(y_test, y_test_pred)) plt.colorbar() plt.xlabel("Predicted label") plt.ylabel("True label")
もう一つの便利な関数はclassification_report()。これは、各クラスの適合率(precision)、再現率(recall)、F1スコア(F1-score)、サポートを表形式で表示してくれる。
from sklearn.metrics import classification_report print classification_report(y_test, y_test_pred)
precision recall f1-score support
0 1.00 1.00 1.00 53
1 0.89 0.91 0.90 43
2 1.00 0.97 0.99 40
3 1.00 0.94 0.97 47
4 0.95 0.98 0.96 41
5 0.96 1.00 0.98 47
6 1.00 0.98 0.99 46
7 1.00 0.96 0.98 52
8 0.85 0.89 0.87 44
9 0.84 0.86 0.85 37
avg / total 0.95 0.95 0.95 450
精度が役立たない状況として不均衡データ(imbalanced data)の例が挙げられている。不均衡データとはたとえば正例が10%で残りの90%が負例のように各クラスのサンプル数が非常に不均衡なデータ。こういうデータだとすべて負例と予測するだけで精度が90%になってしまう。
from sklearn.cross_validation import cross_val_score from sklearn.svm import SVC X, y = digits.data, digits.target == 3 print np.mean(cross_val_score(SVC(), X, y, cv=10))
この例では、数字の3が正例でそれ以外の数字が負例という二値分類問題をSVMで学習し、10-fold CVで評価している。予想通り精度は90%になる。すべてのテストデータに対して「3ではない」と推定するだけでよい。精度は役に立たない。昔これではまったことがあるのを思い出した(笑)
こういうときはROC曲線(ROC curve)とAUC(area under the curve)を使うとより正確に評価できる。ROC曲線は下のコードで描ける。
from sklearn.metrics import roc_curve, roc_auc_score X, y = digits.data, digits.target == 3 X_train, X_test, y_train, y_test = train_test_split(X, y) for gamma in [0.01, 0.05, 1]: plt.xlabel("False Positive Rate") plt.ylabel("True Positive Rate") svm = SVC(gamma=gamma).fit(X_train, y_train) decision_function = svm.decision_function(X_test) fpr, tpr, _ = roc_curve(y_test, decision_function) acc = svm.score(X_test, y_test) auc = roc_auc_score(y_test, svm.decision_function(X_test)) plt.plot(fpr, tpr, label="acc:%0.2f auc:%0.2f" % (acc, auc), linewidth=3) plt.legend(loc="best")
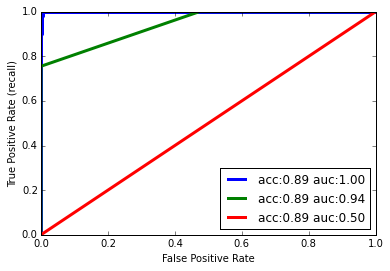
ROC曲線は横軸にFalse Positive Rate、縦軸にTrue Positive Rate(Recall)を割り当て、さまざまな閾値でこの2つ組をプロットしたグラフである。
横軸のFalse Positive Rate(FPR)は、本来は負例なのに誤って(False)正例(Positive)と判定してしまった割合なので小さいほどよい。一方、縦軸のTrue Positive Rate(TPR)は、正しく(True)正例(Positive)と判定できた割合なので大きい方がよい。つまり、カーブが左上に行くほど分類器の性能がよいことになる。このカーブの下の面積がAUCで1.0のとき最良となる。
この結果を見るとSVMのパラメータ(gamma)をいろいろ変えても先と同じように精度(accuracy)はすべて90%になってしまい優劣の比較ができない。しかし、ROC曲線とAUCの値はパラメータによって異なっており、gamma = 0.01(青の曲線)がもっともよいと判定できる。つまり、精度で評価するよりAUCで評価した方がよい場合がある。
ROC曲線は名前だけ聞いたことあったけどほとんど使ったことなかった。このページの説明はわかりやすいと思った。あとは朱鷺の杜のROC曲線のページも。
sklearnには上記以外にもさまざまなスコアがすでに実装されている。
from sklearn.metrics.scorer import SCORERS print SCORERS.keys()
['f1', 'f1_weighted', 'f1_samples', 'recall_samples', 'recall_micro', 'adjusted_rand_score', 'recall_macro', 'mean_absolute_error', 'precision_macro', 'precision_weighted', 'f1_macro', 'recall_weighted', 'accuracy', 'precision_samples', 'median_absolute_error', 'precision', 'log_loss', 'precision_micro', 'average_precision', 'roc_auc', 'r2', 'recall', 'mean_squared_error', 'f1_micro']
mean_squared_errorのようなよく使うものもあるけど、聞いたことないのもいろいろあるんだな。
ここまでで4章終わり。モデル評価は地味な感じがするけど、機械学習の応用では必須の知識になりそう。5章からはまた内容が一転してさまざまな分類器の詳細な説明が始まっている。長くなったのでつづきはPart IIIで。