KerasでMNIST
今回は、KerasでMNISTの数字認識をするプログラムを書いた。このタスクは、Kerasの例題にも含まれている。今まで使ってこなかったモデルの可視化、Early-stoppingによる収束判定、学習履歴のプロットなども取り上げてみた。
ソースコード: mnist.py
MNISTデータのロードと前処理
MNISTをロードするモジュールはKerasで提供されているので使った。
from keras.datasets import mnist from keras.utils import np_utils # MNISTデータのロード (X_train, y_train), (X_test, y_test) = mnist.load_data() # 画像を1次元配列化 X_train = X_train.reshape(60000, 784) X_test = X_test.reshape(10000, 784) # 画素を0.0-1.0の範囲に変換 X_train = X_train.astype('float32') X_test = X_test.astype('float32') X_train /= 255 X_test /= 255 print(X_train.shape[0], 'train samples') print(X_test.shape[0], 'test samples') # one-hot-encoding Y_train = np_utils.to_categorical(y_train, nb_classes) Y_test = np_utils.to_categorical(y_test, nb_classes)
KerasでダウンロードしたMNISTのデフォルトの形状は (60000, 28, 28) なので (60000, 784) に reshapeする。各サンプルが784次元ベクトルになるようにしている。画像データ(X)は0-255の画素値が入っているため0.0-1.0に正規化する。クラスラベル(y)は数字の0-9が入っているためone-hotエンコーディング型式に変換する。
nb_classes = 10を省略するとy_testに入っているラベルから自動的にクラス数を推定してくれるようだが、必ずしも0-9のラベルすべてがy_testに含まれるとは限らないためnb_classes = 10を指定したほうが安全のようだ。
モデルの可視化
from keras.models import Sequential from keras.layers.core import Dense, Dropout, Activation from keras.optimizers import Adam from keras.utils.visualize_util import plot def build_multilayer_perceptron(): model = Sequential() model.add(Dense(512, input_shape=(784,))) model.add(Activation('relu')) model.add(Dropout(0.2)) model.add(Dense(512)) model.add(Activation('relu')) model.add(Dropout(0.2)) model.add(Dense(10)) model.add(Activation('softmax')) return model # 多層ニューラルネットワークモデルを構築 model = build_multilayer_perceptron() # モデルのサマリを表示 model.summary() plot(model, show_shapes=True, show_layer_names=True, to_file='model.png') # モデルをコンパイル model.compile(loss='categorical_crossentropy', optimizer=Adam(), metrics=['accuracy'])
隠れ層が2つある多層パーセプトロンを構築した。活性化関数には relu。また、過学習を防止するテクニックである Dropout を用いた。Dropoutも層として追加する。
model.summary()を使うと以下のようなモデル形状のサマリが表示される。modelにadd()で追加した順になっていることがわかる。Output ShapeのNoneはサンプル数を表している。dense_1 層のパラメータ数(重み行列のサイズのこと)は となる。512を足すのはバイアスも重みに含めているため。ユーザがバイアスの存在を気にする必要はないが、裏ではバイアスも考慮されていることがパラメータ数からわかる。同様に
dense_2 層のパラメータ数は となる。同様に
dense_3 層のパラメータ数は となる。
____________________________________________________________________________________________________ Layer (type) Output Shape Param # Connected to ==================================================================================================== dense_1 (Dense) (None, 512) 401920 dense_input_1[0][0] ____________________________________________________________________________________________________ activation_1 (Activation) (None, 512) 0 dense_1[0][0] ____________________________________________________________________________________________________ dropout_1 (Dropout) (None, 512) 0 activation_1[0][0] ____________________________________________________________________________________________________ dense_2 (Dense) (None, 512) 262656 dropout_1[0][0] ____________________________________________________________________________________________________ activation_2 (Activation) (None, 512) 0 dense_2[0][0] ____________________________________________________________________________________________________ dropout_2 (Dropout) (None, 512) 0 activation_2[0][0] ____________________________________________________________________________________________________ dense_3 (Dense) (None, 10) 5130 dropout_2[0][0] ____________________________________________________________________________________________________ activation_3 (Activation) (None, 10) 0 dense_3[0][0] ==================================================================================================== Total params: 669706
keras.utils.visualize_util の plot() を使うとモデルを画像として保存できる。今はまだ単純なモデルなので summary() と同じでありがたみがないがもっと複雑なモデルだと図の方がわかりやすそう。

Early-stoppingによる収束判定
これまでの実験では、適当に nb_epoch を決めて固定の回数だけ訓練ループを回していたが過学習の危険がある。たとえば、このMNISTタスクで100回回したときの loss(訓練データの損失)と val_loss(バリデーションセットの損失)をプロットすると下のようになる(次節のhistoryでプロット)。

この図からわかるように loss はエポックが経つにつれてどんどん下がるが、逆に val_loss が上がっていくことがわかる。これは、訓練データセットに過剰にフィットしてしまうために未知のデータセットに対する予測性能が下がってしまう過学習を起こしていることを意味する。機械学習の目的は未知のデータセットに対する予測性能を上げることなので過学習はダメ!
普通は訓練ループを回すほど性能が上がりそうだけど、先に見たように訓練ループを回せば回すほど性能が悪化する場合がある。そのため、予測性能が下がる前にループを打ち切りたい。val_loss をプロットして目視でどこで打ち切るか判断することもできるが、それを自動で判断してくれるのがEarly-stoppingというアルゴリズム。
If the model’s performance ceases to improve sufficiently on the validation set, or even degrades with further optimization, then the heuristic implemented here gives up on much further optimization. http://deeplearning.net/tutorial/gettingstarted.html#early-stopping
Theanoでは自分で実装(2015/5/26)したが、Kerasではコールバック関数としてEarlyStoppingが実装されているためfit()のcallbacksオプションに設定するだけでよい(うまい実装だね~)。EarlyStoppingを使うには必ずバリデーションデータセットを用意する必要がある。fit()のオプションでvalidation_dataを直接指定することもできるが、validation_splitを指定することで訓練データの一部をバリデーションデータセットとして使える。
Keras examplesもそうだが、テストデータセットをバリデーションデータセットとして使うのは本来ダメらしい。バリデーションデータセットとテストデータセットは分けたほうがよい。何となく直感的にダメそうというのはわかるのだがどうしてかは実はよく知らない。
# Early-stopping early_stopping = EarlyStopping(patience=0, verbose=1) # モデルの訓練 history = model.fit(X_train, Y_train, batch_size=batch_size, nb_epoch=nb_epoch, verbose=1, validation_split=0.1, callbacks=[early_stopping])
EarlyStoppingを導入するとわずか5エポックくらいで訓練が打ち切られる。確かにここら辺からval_lossが上がってくるのでよいのかもしれないが少し早すぎかもしれない。EarlyStoppingにはpatienceというパラメータを指定できる。デフォルトでは0だが、patienceを上げていくと訓練を打ち切るのを様子見するようになる。通常はデフォルトでOKか?
verboseが機能していない?どこに表示されるのだろう?
学習履歴のプロット
fit()の戻り値である history に学習経過の履歴が格納されている。このオブジェクトを使えばいろいろな経過情報をプロットできる。デフォルトでは、loss(訓練データセットの損失)だけだが、modelのmetricsにaccuracyを追加するとacc(精度)が、バリデーションデータセットを使うとval_loss(バリデーションデータセットの損失)やval_acc(バリデーションデータセットの精度)が自動的に追加される。さっきの図はこのデータを使ってmatplotlibで書いた。ユーザ独自のメトリクスも定義できるようだ。
def plot_history(history): # print(history.history.keys()) # 精度の履歴をプロット plt.plot(history.history['acc']) plt.plot(history.history['val_acc']) plt.title('model accuracy') plt.xlabel('epoch') plt.ylabel('accuracy') plt.legend(['acc', 'val_acc'], loc='lower right') plt.show() # 損失の履歴をプロット plt.plot(history.history['loss']) plt.plot(history.history['val_loss']) plt.title('model loss') plt.xlabel('epoch') plt.ylabel('loss') plt.legend(['loss', 'val_loss'], loc='lower right') plt.show() # 学習履歴をプロット plot_history(history)
今回は、Kerasの便利なツールをいろいろ使ってみた。
参考
Kerasによる多クラス分類(Iris)
今回は、機械学習でよく使われるIrisデータセットを多層パーセプトロンで分類してみた(ありがち)。Irisデータセットのクラスラベルは3つ(setosa, versicolor, virginica)あるので前回までと違って多クラス分類になる。短いプログラムなので全部載せてポイントだけまとめておこう。

ソースコード
import numpy as np from sklearn import datasets from sklearn.model_selection import train_test_split from keras.models import Sequential from keras.layers.core import Dense, Activation from keras.utils import np_utils from sklearn import preprocessing def build_multilayer_perceptron(): """多層パーセプトロンモデルを構築""" model = Sequential() model.add(Dense(16, input_shape=(4, ))) model.add(Activation('relu')) model.add(Dense(3)) model.add(Activation('softmax')) return model if __name__ == "__main__": # Irisデータをロード iris = datasets.load_iris() X = iris.data Y = iris.target # データの標準化 X = preprocessing.scale(X) # ラベルをone-hot-encoding形式に変換 # 0 => [1, 0, 0] # 1 => [0, 1, 0] # 2 => [0, 0, 1] Y = np_utils.to_categorical(Y) # 訓練データとテストデータに分割 train_X, test_X, train_Y, test_Y = train_test_split(X, Y, train_size=0.8) print(train_X.shape, test_X.shape, train_Y.shape, test_Y.shape) # モデル構築 model = build_multilayer_perceptron() model.compile(optimizer='adam', loss='categorical_crossentropy', metrics=['accuracy']) # モデル訓練 model.fit(train_X, train_Y, nb_epoch=50, batch_size=1, verbose=1) # モデル評価 loss, accuracy = model.evaluate(test_X, test_Y, verbose=0) print("Accuracy = {:.2f}".format(accuracy))
ポイント
irisデータは
sklearn.datasets.load_iris()でダウンロードできる。よく使う標準データセットはメソッドが用意されていて便利irisのラベルは文字列だがsklearnのデータセットでは、0, 1, 2のように数値ラベルに変換されている。これをニューラルネットで扱いやすいone-hotエンコーディング型式に変換する。上のコメントにも書いたようにone-hotエンコーディングは、特定のユニットのみ1でそれ以外は0のようなフォーマットのこと。この変換は、
keras.utils.np_utilsのto_categorical()に実装されている訓練データとテストデータは、
sklearn.model_selectionのtrain_test_split()を使う。訓練データが8割、テストデータが2割になるように分割した。少し古いscikit-learnだとsklearn.cross_validationに実装されているけどこのモジュールはすでにdeprecated。モデル構築は
build_multilayer_perceptron()という独自関数を用意した。モデルが複雑になると関数化したほうがわかりやすそう。入力層が4ユニット、隠れ層が16ユニット、出力層が3ユニットの多層パーセプトロンを構築した多クラス分類の場合は、損失関数に
categorical_crossentropyを指定する
必要な前処理メソッドはたいていscikit-learnに実装されているので自分で実装する前に探してみたほうがよさそう。
5回くらい実行してテストデータの精度を求めると97%、93%、100%、93%、97%となった。どのサンプルがテストデータに選ばれるかによって精度が変わるようだ。分類境界あたりのサンプルがテストデータとして選ばれると予測が難しくなり精度が下がりそうなのは直感的にわかる。
こういう場合は、何度か実行して平均を求めるのがセオリーかな。でも巨大なデータセットの学習だと何日もかかるケースがありそうだし、そんなに何回もデータセットの分割を変えて実行はできないなあ。どうするのが一般的なんだろうか?
参考
Kerasによる2クラス分類(Pima Indians Diabetes)
Kerasのプログラミングは
- データのロード
- モデルの定義
- モデルのコンパイル
- モデルの学習
- モデルの評価
- 新データに対する予測
という手順が一般的。今回はもう少し実践的なデータを使ってこの流れをつかみたい。
ソースコード:pima.py
1. データのロード
参考文献で挙げた記事と同じようにUCI Machine Learning repositoryにあるPima Indians Diabetes Data Setを使おう。 医療系のデータでPimaインディアンが糖尿病にかかったかどうかを表すデータのようだ。
Attribute Information: 1. Number of times pregnant 2. Plasma glucose concentration a 2 hours in an oral glucose tolerance test 3. Diastolic blood pressure (mm Hg) 4. Triceps skin fold thickness (mm) 5. 2-Hour serum insulin (mu U/ml) 6. Body mass index (weight in kg/(height in m)^2) 7. Diabetes pedigree function 8. Age (years) 9. Class variable (0 or 1)
データの説明によると最初の8列が患者の属性情報で9列目が糖尿病を発症しない(0)または糖尿病を発症した(1)というラベルとなっている。つまり、患者の属性情報から糖尿病を発症するかを予測するモデルを学習するのがここでの目的。
このデータはCSV形式でダウンロードできる。データをロードするコードは、
# load pima indians dataset dataset = np.loadtxt(os.path.join("data", "pima-indians-diabetes.data"), delimiter=',') # split into input (X) and output (Y) variables X = dataset[:, 0:8] Y = dataset[:, 8]
ここで、二次元データならグラフで可視化してみるところだけれど、8次元データはそのままでは可視化できない。分析するなら属性間の相関図や次元圧縮してみるのがセオリーか?今回はKerasの使い方の習得がメインなので飛ばそう。
2. モデルの定義
ここはニューラルネットの構造を組み立てるところ。今回は、隠れ層が2つ、出力層が1つの多層パーセプトロン を構築する。
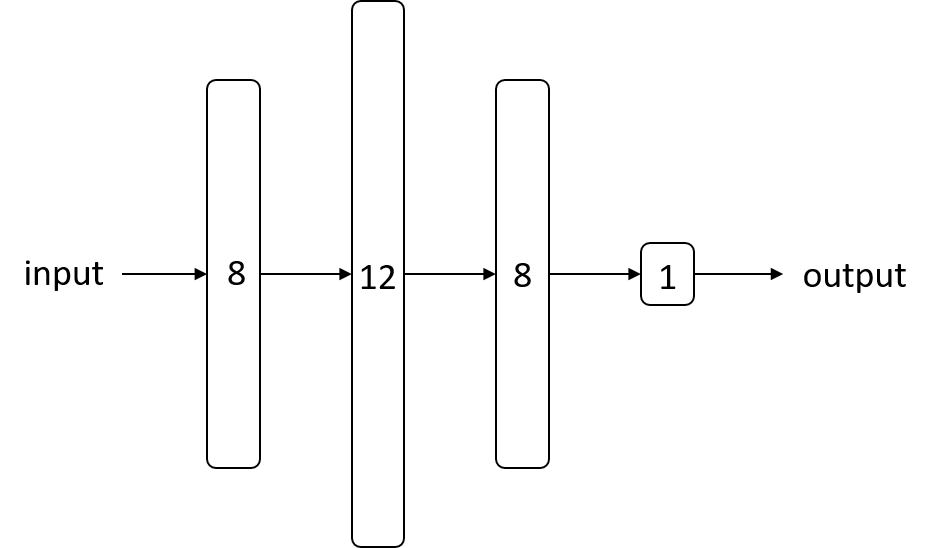
# create model model = Sequential() model.add(Dense(12, input_dim=8, init='uniform', activation='relu')) model.add(Dense(8, init='uniform', activation='relu')) model.add(Dense(1, init='uniform', activation='sigmoid'))
initで層の重みの初期化方法を指定できるuniformだと0~0.05の一様乱数。normalだと正規乱数。Deep Learning Tutorialの初期値重みで使われていたglorot_uniformもある- 層の活性化関数は、独立した層ではなく
Denseのactivation引数でも指定できる - 隠れ層の活性化関数には
relu、出力層の活性化関数にはsigmoidを指定 - 出力層に
sigmoidを使うと0.0から1.0の値が出力されるため入力データのクラスが1である確率とみなせる。0.5を閾値として0.5未満ならクラス0、0.5以上ならクラス1として分類する
3. モデルのコンパイル
損失関数、最適化関数、評価指標を指定してモデルをコンパイルする。2クラス分類なのでbinary_crossentropyを使う。ここら辺は前回と同じ。
# compile model model.compile(loss='binary_crossentropy', optimizer='adam', metrics=['accuracy'])
4. モデルの学習
# fit the model model.fit(X, Y, nb_epoch=150, batch_size=10)
- 訓練データ
XとラベルYを指定して学習 - エポックは固定で150回ループを回す
- 学習はいわゆるミニバッチ学習でバッチサイズは10とした。データを10個ずつ取り出して誤差を蓄積し、その誤差で1回重みを更新する。
5. モデルの評価
# evaluate the model scores = model.evaluate(X, Y) print("%s: %.2f%%" % (model.metrics_names[1], scores[1] * 100))
- モデルの評価には
model.evaluate()を使う。 - 戻り値は評価指標のリスト。デフォルトでは損失(loss)のみ。
compile()のmetricsに評価指標を追加すると、別の評価指標が追加される。今回は、精度(acc)を追加してある。model.metrics_namesで、評価尺度の名前リストが得られる。
> print(model.metrics_names) ['loss', 'acc'] > print(scores) [0.45379118372996646, 0.7890625]
ここでは、訓練データを使って評価しているが、実際は訓練データとは独立したテストデータを用意するべき。ただ、モデルにバグがないか確認するために最初は訓練データで評価してみるのもありだと思う。訓練データでさえ精度がものすごく低かったらそもそも学習できないかプログラムにバグがある。今回は、訓練データの予測で精度79%なのでそこそこうまくいっている?次回は訓練データとテストデータを分ける例を取り上げたい。
6. 新データに対する予測
predictions = np.round(model.predict(X))
correct = Y[:, np.newaxis]
- モデルに新しいデータを入力してクラスラベルを予測するには
model.predict() - 今回は簡単のため訓練データ
Xをそのまま入力 - 出力層は
sigmoidなので下のように0.0から1.0の値が出力される - 出力をクラスラベルにするために
round()を使う。0.5以上なら1に切り上げ、未満なら0に切り下げ
> print(predictions) [[ 0.82117373] [ 0.10473501] [ 0.90901828] [ 0.10512047] [ 0.78090101] ...
> print(correct) [[ 1.] [ 0.] [ 1.] [ 0.] [ 1.] ...
今回は実践的なデータセットを用いてKerasによる実験の流れを一通りまとめた。